Neurogenetik
Binäre Expressionssysteme
Um ein bestimmtes Gen von Interesse in einem spezifischen Zell-/Gewebsmuster zu exprimieren, verwenden wir komplementäre binäre Expressionssysteme - GAL4-UAS (obere linke Grafik) und lexA-lexAop (obere rechte Grafik) - die Transaktivatoren und Responder-Elemente separat kodieren. Eine Driver-Fliegenlinie exprimiert das Hefe-Transkriptionsaktivatorprotein GAL4 (oberes linkes Diagramm) oder ein chimäres Transkriptionsaktivatorprotein, das aus dem bakteriellen Repressor LexA besteht, der mit der C-terminalen Aktivierungsdomäne von GAL4 (oberes rechtes Diagramm) fusioniert ist, unter der Kontrolle eines spezifischen Promotors/Enhancers. Eine Responder-Fliegenlinie trägt ein Gen von Interesse (Gen X) unter UAS (Upstream Activation Sequence, obere linke Grafik), einem Enhancer, an den GAL4 spezifisch bindet, um die Gentranskription zu aktivieren, oder unter einem genspezifischen LexA-DNA-Bindungsmotiv (LexAop, obere rechte Grafik). Wenn eine Treiberlinie mit einer entsprechenden Responderlinie gekreuzt wird, exprimiert die Nachkommenschaft das interessierende Gen (Gen X) unter der Regulation des spezifischen Promotors/Enhancers. Ein dritter Typ von Treiberfliegenlinien (untere Grafik), die GAL80ts, einen temperatursensitiven GAL4-Repressor aus Hefe, exprimieren, bietet eine zeitliche Auflösung des GAL4-UAS-Systems. Unter der restriktiven Temperatur (18°C) bindet GAL80 an die Transkriptionsaktivierungsdomäne von GAL4 und blockiert dessen Aktivität; wenn die Fliegen auf die permissive Temperatur (29°C) transferiert werden, setzt GAL80 GAL4 frei, um die Genexpression über UAS zu aktivieren.

Transcriptomics / RNAseq
Genfamilien, die an der Erkennung von Geruchsstoffen beteiligt sind, weisen eine enorme Sequenzvariabilität und bei den meisten Arten eine große Anzahl von Genen auf. Unser Labor hat vor einigen Jahren mit dem Einsatz der transkriptomischen Sequenzierung begonnen, um die Identifizierung und Untersuchung der Evolution der entsprechenden Genfamilien zu ermöglichen, insbesondere bei Arten, die aus Sicht der Evolutionsbiologie relevant sind, aber keine Modellarten mit existierenden Datensätzen sind.

Deorphanisierung von Rezeptoren auf der Grundlage von Expressionsänderungen der mRNA-Spiegel (DREAM)
Ein wichtiger erster Schritt für viele neurogenetische Ansätze im Geruchssinn ist die Assoziation von Geruchsstoffen und detektierenden Rezeptoren. Die 2015 von von der Weid et al. veröffentlichte DREAM-Methode nutzt zu diesem Zweck den Nachweis der Rezeptor-mRNA-Regulation nach längerer Geruchsstimulation. Wir haben die Anwendung von DREAM etabliert und detektieren rezeptorkodierende Transkripte mit qPCR, RNAseq oder Nanostring-Technologie. Ein wesentliches Merkmal der Methode ist die Konzentration auf den angewandten Geruch - wir stellen nicht die Frage "Was macht dieser Rezeptor?", sondern "Welche Rezeptoren sind für die Erkennung dieses verhaltensmäßig/ökologisch relevanten Geruchs wichtig?".

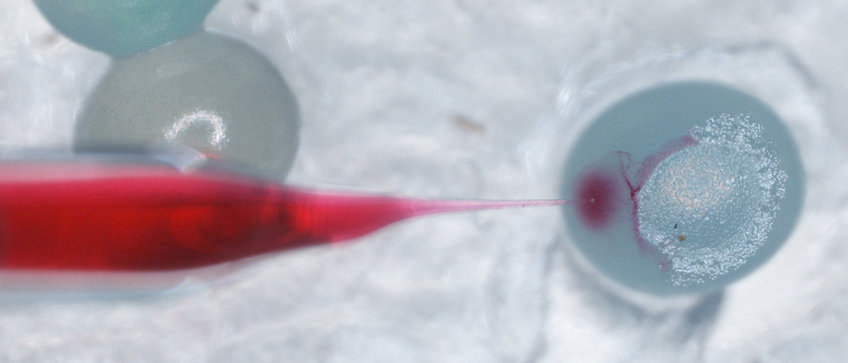
CRISPR Cas9
Die gezielte Genbearbeitung in vivo mit CRISPR Cas9 hat in den letzten Jahren für Schlagzeilen gesorgt und den Bereich der Genmanipulation revolutioniert. Wir haben die Methode für den Einsatz beim Tabakhornwurm Manduc sexta etabliert, wodurch wir eine Verbindung zwischen Genen, Genfunktionen und Verhalten herstellen können.




