
Neuroanatomie
Photoactivation

Um Neuronen auf der Ebene einzelner Zellen zu analysieren, verwenden wir ein genetisch kodiertes photoaktivierbares GFP (PA-GFP) zur neuronalen Verfolgung. PA-GFP wird bei einer kontinuierlichen Beleuchtung von 760 nm photokonvertiert, was zu einer 100-fachen Steigerung der GFP-Fluoreszenz führt. Durch Photoaktivierung einzelner Somata mit einem IR-Laser am 2-Photonen-Mikroskop können einzelne Neuronen selektiv von ihrem Soma bis zu ihren entferntesten axonalen Endigungen markiert werden. Danach wird ein z-Stapel des gesamten Gehirns aufgenommen und anschließend für neuronale 3D-Rekonstruktionen verwendet. Alle rekonstruierten Neuronen werden in unser Standard-in-vivo-Gehirn transformiert und registriert, um die Neuronen verschiedener Individuen anzugleichen.
Neuronal Tracing
Wir setzen computergestützte, halbautomatische digitale Techniken ein, um neuronale Merkmale und Schaltkreise im Gehirn zu kartieren. Bilddaten werden durch konfokales Laserscanning (CSLM), Elektronenmikroskopie (TEM, FIB-SEM) sowie neuronale Tracings in Korrelation mit physiologischen Experimenten (optische Bildgebung, Elektrophysiologie und Patch-Clamp) gewonnen. Mit Hilfe der 3D-Rekonstruktionssoftware Amira-Software verfolgen wir halbautomatisch die Neuronenmorphologie und segmentieren die entsprechenden Hirnregionen (Neuropillen). Die quantitative Morphometrie ermöglicht uns die statistische Analyse gängiger Neuronenparameter. In Kombination mit Bildregistrierungstechniken können morphologische Daten mit physiologischen und molekularen Daten verknüpft und artübergreifend verglichen werden.

Image Registration
Die Bildregistrierung ist eine häufig verwendete Technik, um neuronale Morphologien in einem gemeinsamen 3D-Referenzraum zu visualisieren. Bei diesem Koordinatentransformationsprozess werden die Koordinaten eines Bildes auf den anatomisch äquivalenten Punkt in einem anderen Bild abgebildet. Wir verwenden Grauwert-, Labelfeld- und Landmarken-basierte Registrierung mit der Software Amira (FEI?). Die Registrierung ermöglicht es uns schließlich, Daten über verschiedene Modalitäten und Experimente hinweg in einem gemeinsamen Referenzsystem zu analysieren und zu vergleichen (Rybak, 2016).

GRASP
Wir verwenden synaptisch getaggte GRASP-Konstrukte, bei denen spGFP1-10 an Neurexin (ein für synaptische Stellen spezifisches Transmembranprotein) und spGFP11 an die Membran fusioniert ist. Wir verwenden zwei verschiedene binäre Transkriptionssysteme: Gal4-UAS und LexA-LexAop, um jede GFP-Fraktion in zwei verschiedenen neuronalen Populationen zu exprimieren.
Das GRASP-Signal wird in vitro nach Immunolabeling mit lichtmikroskopischen Techniken sichtbar gemacht.

Electron Microscopy
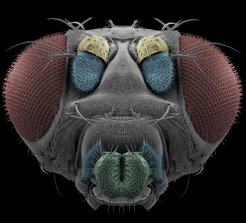
Die Rasterelektronenmikroskopie (REM) wird eingesetzt, um Kutikula-Strukturen, wie z. B. sensorische Sensillen, bei verschiedenen Insektenarten (Manduca sexta, Drosophila melanogaster und Crustacea) sichtbar zu machen und zu quantifizieren. Mit dieser Technik können äußere morphologische Merkmale mit hoher Auflösung dargestellt werden.
Die klassische Transmissionselektronenmikroskopie (TEM) wird in Verbindung mit der konfokalen Mikroskopie von genetisch markierten Neuronen eingesetzt, um die synaptische Konnektivität in Drosophila melanogaster zu untersuchen. Ultradünne Schnitte werden manuell geschnitten, auf Gittern gesammelt und dann im TEM fotografiert. Serielle, aufeinanderfolgende Schnitte werden mit der Open-Source-Software Trak-EM (Image J-Fiji) ausgerichtet und neuronale Profile segmentiert. 3D-Rendering-Techniken ermöglichen die kollektive Visualisierung synaptischer Netzwerke im 3D-PDF-Format. (Rybak et al., 2016).
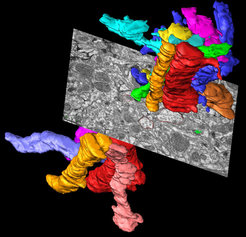
Darüber hinaus setzen wir automatisierte EM-Techniken in großem Maßstab ein, wie z. B. die Focused Ion Beam-Scanning Electron Microscopy (FIB-SEM), um vollständige und schnelle Rekonstruktionen neuronaler Netzwerke auf ultrastruktureller Ebene zu erhalten. Laser-Branding (unter Verwendung der Zwei-Photonen-Laser-Scanning-Mikroskopie) wird verwendet, um die Hirnregion von Interesse auf ultrastruktureller Ebene zu identifizieren. Für die Datenerfassung wird Open-Source-Software wie CatMaid http://openconnecto.me/catmaid/ verwendet.
LightSheet Microscopy

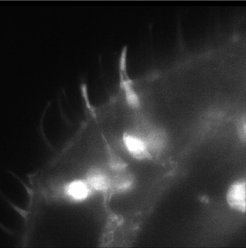
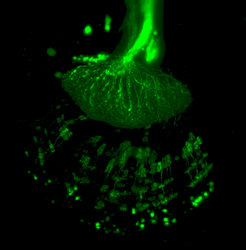
Die Lichtblatt-Fluoreszenzmikroskopie (Light Sheet Fluorescence Microscopy, LSFM) ist ein 3D-Tomographieverfahren, bei dem im Gegensatz zur punktuellen Laserabtastung der konfokalen Mikroskopie ein dünnes Lichtblatt verwendet wird. Vorteile sind schnelle Scanzeiten und hoher Kontrast, die hochauflösende Scans in Raum und Zeit ermöglichen. Das schnelle Scannen reduziert Fotoschäden und ist daher am besten für die in-vivo-Bildgebung von fluoreszenzmarkierten Strukturen geeignet.
Derzeit setzen wir diese Technik ein, um schnelle Scans des gesamten Gehirns und der äußeren Kutikula bei verschiedenen Insektenarten zu erhalten.








