Mit der massenspektrometrischen Lupe auf der Suche nach Stoffwechselprodukten
Forschungsbericht (importiert) 2010 - Max-Planck-Institut für chemische Ökologie
Einführung
Um ein Stoffwechselprodukt direkt im Gewebe lokalisieren zu können, haben Wissenschaftler kürzlich neue Methoden der Gewebe- und Zellvisualisierung entwickelt. Diese Techniken bestehen aus der Beobachtung Molekül-spezifischer Vibrationen durch Spektroskopie, wie zum Beispiel NMR, nuclear magnetic resonance, und CARS, Coherent anti-Stokes Raman scattering [1], oder aus direkter Messung der Masse der Moleküle innerhalb der biologischen Probe durch Massenspektrometrie (MS). In Kombination mit Farb- oder anderen Markierungsmöglichkeiten stellen diese neuen Methoden eine weitere Dimension bei Untersuchungen biologischen Materials mit bildgebenden Verfahren dar, denn sie sind weniger invasiv und erlauben einen direkten Einblick in das Zellgeschehen.
Die Massenspektrometrie ist traditionell ein analytisches Verfahren zur Messung der Masse von ionisierten Molekülen. Verwendet werden Massenspektrometer, das sind Instrumente, die im Wesentlichen aus drei Teilen bestehen: einer Messquelle, einem Massenanalysator und einem Detektor. In der Messquelle befindet sich die zu analysierende Probe, die in einen gasförmigen Zustand versetzt, ionisiert und nachfolgend einem Massenanalysator zugeführt wird. Dort werden die eintreffenden Ionen unter hohem Vakuum je nach Masse-zu-Ladung-Verhältnis (m/z) aufgetrennt und anschließend detektiert. Alle Teile des Massenspektrometers sind mit Computern verbunden, die die Hardware kontrollieren und auch die Signale des Detektors aufzeichnen. Das Ergebnis – das sogenannte Massenspektrum – zeigt die Intensität der detektierten Ionen im Verhältnis zu ihrer Masse-zu-Ladung-Relation (m/z).
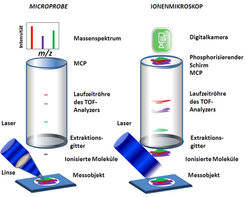
Massenspektrometrisches Imaging (MSI) kann in zwei Haupttypen aufgeteilt werden: Scanning (Microprobe) und Ionenmikroskopie (Abb. 1). In der ersten, weiter verbreiteten Methode werden die Moleküle von Interesse innerhalb eines zuvor auf der biologischen Probe umrissenen Bereiches (definiert in Pixel oder Voxel) durch Laserstrahl, Teilchenstrahl oder jetstream desorbiert und ionisiert. Nachfolgend kommen Massenanalysatoren und -detektoren zum Einsatz, die das Masse-zu-Ladung-Verhältnis (m/z), die Intensität und möglicherweise auch die Struktur der detektierten Moleküle mittels „Tandem-Massenspektrometrie“, das heißt in Kombination mit collision-induced-dissociation (CID), ermitteln. Dadurch erhält man Massenspektren in den ausgewählten Masse-zu-Ladung-Bereichen von Hunderten bis Tausenden von Punkten der zuvor definierten Koordinaten auf der biologischen Probe, vergleichbar der Methodik bei der Rasterelektronenmikroskopie. Nachdem einzelne Peaks desorbierter Ionen in den jeweiligen Koordinationspunkten per Computer verzeichnet wurden, erhält man zwei- oder sogar dreidimensionale Darstellungen der Intensitäten, normalerweise als Falschfarbenbilder. Diese können virtuell auf ein herkömmliches Foto der gemessenen biologischen Probe, beispielsweise ein Pflanzenblatt, projiziert werden. Zusätzlich kann eine individuelle „Intensitätskarte“ für jedes Massenmerkmal oder jede einzelne chemische Einheit erstellt werden. Auf diese Weise können unverfälschte metabolomische Untersuchungen durchgeführt werden, beispielsweise von Probenmaterial, das wechselnden Umweltbedingungen ausgesetzt war und in der Folge unterschiedlich regulierte Stoffwechselaktivitäten aufweist. Dies macht die Methode des massenspektrometischen Imaging attraktiv für Anwendungen besonders in der chemischen Ökologie [2].
MSI-Spektren von intaktem Gewebe und anderen Objekten
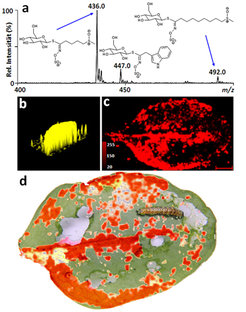
Wissenschaftler aus der Abteilung Biochemie des Max-Planck-Instituts für chemische Ökologie hatten beobachtet, dass Raupen beim Fressen von Blättern der Ackerschmalwand (Arabidopsis thaliana) deren Ränder sowie die Mittelrippe mieden. Bekannt war auch, dass die Ackerschmalwand von Aminosäuren abgeleitete Pro-Phytoalexine, sogenannte Glucosinolate, enthält, die die Pflanze als Gift gegen Fraßfeinde einsetzt [3]. Die Kenntnis über deren genaue Verteilung auf den Blättern könnte vielleicht das Fraßverhalten der Insekten erklären. Daher wollten sich die Biochemiker der MSI-Technik bedienen, zumal es den Forschern bislang nicht gelungen war, die Verteilung bestimmter Sekundärmetabolite in intaktem Pflanzengewebe auf feinster, quasi zellulärer Skala zu bestimmen. Tatsächlich konnte eine erstaunlich einheitliche Verteilung von Glucosinolaten in den Blättern festgestellt werden, wobei im Vergleich zum übrigen Blatt eine größere Menge in der Mittelrippe und dem Blattrand auftritt [4], (Abb. 2). Es wird vermutet, dass die höhere Glucosinolat-Konzentration am Blattrand eine Art Schutzwall gegen kleine Fraßinsekten darstellt und dass auf diese Weise eine effektivere Abwehr gewährleistet wird als bei einer gleichmäßigen Verteilung der Glucosinolate im gesamten Blattgewebe. Diese Annahme wurde nachfolgend durch Bioassays des Fraßverhaltens der Insekten und durch andere Vergleichsexperimente, wie zum Beispiel durch Bestimmung der jeweiligen Glucosinolatkonzentrationen in einzelnen Blattabschnitten mittels Flüssigchromatografie oder durch gezielte Verwendung von A. thaliana-Mutanten, bestätigt. Die charakteristische Verteilung von Glucosinolaten in den Blättern der Ackerschmalwand scheint demnach tatsächlich die Abwehrfunktion in ausgeklügelter und unerwarteter Weise zu verstärken. Ohne die in diesem Artikel vorgestellte deutliche Verbesserung bereits existierender Imaging-Methoden, die erst die Analyse von Stoffwechselprodukten wie Glucosinolaten in dem kleinen Mengenbereich ermöglicht hat, der für kleine pflanzenfressende Insekten bereits relevant ist, wären solche interessanten Rückschlüsse auf pflanzliche Verteidigungsstrategien nicht möglich gewesen.
Dieser Erfolg hat zu einer Vielzahl von Studien geführt, in denen die Verteilung von Naturstoffen in bildgebenden Verfahren dargestellt wird, wobei die analysierten Stoffe von unpolaren Kohlenwasserstoffen [5] bis hin zu größeren Antibiotika-Molekülen reichen, die beispielsweise von symbiotisch lebenden Bakterien in den Antennen weiblicher Bienenwölfe – einer Wespenart – synthetisiert werden [6]. In letzterer Studie konnten die Antibiotika erstmals direkt in ihrer natürlichen Umgebung, d.h. auf dem Bienenwolfkokon (Abb. 3b), identifiziert werden; dort schützen sie die junge Brut vor bakteriellen Pathogenen und Pilzinfektionen. Damit gelang auch gleichzeitig der Beweis, dass die symbiotischen Bakterien die Antibiotika wirklich in ihrem natürlichen Lebensraum – also dem Kokon – herstellen und nicht nur in isolierter Form auf künstlichen Nährmedien im Labor! In dem antibiotischen Cocktail befand sich neben acht verschiedenen Piericidinen Streptochlorin, ein Chlorin-substituiertes Indol-Alkaloid. Mithilfe einer weiterentwickelten bildgebenden Massenspektrometrie-Technik (Laser-Desorption/Ionisierung, LDI-Imaging, [7]) konnte sogar gezeigt werden, dass die Antibiotika hauptsächlich auf der Außenseite des Kokons vorhanden waren. Dadurch wird das Risiko potenzieller negativer Nebenwirkungen auf die Larve verringert. Darüber hinaus konnten die Wissenschaftler zeigen, dass der Einsatz von verschiedenen Arten von Antibiotika einen sehr effektiven Schutz gegen Infektionen darstellt, die aus einer Vielzahl verschiedener pathogener Mikroorganismen resultieren. Bienenwölfe machen sich also seit Millionen von Jahren ein Prinzip zu Nutze, das in der Humanmedizin als Kombinationsprophylaxe bezeichnet wird.
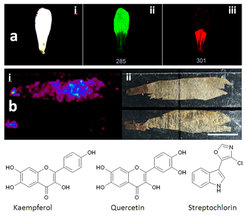
Die LDI-Imaging-Technik war in Zusammenarbeit mit der Abteilung Entomologie des Instituts und dem Institut für Organische Chemie der Friedrich-Schiller-Universität Jena entwickelt worden, um molekulare Karten mit zellulärer Auflösung erstellen zu können [7]. Mit der ursprünglichen Methode, bei der die MALDI-Matrixlösung über die Oberfläche des abzubildenden Gewebes gesprüht wird, ist nämlich das Problem verbunden, dass sich kleine Tröpfchen der Lösung zu größeren verbinden können und die daraus resultierende Ko-Kristallisierung der Matrix mit den zu messenden Metaboliten zur Verfälschung der Ergebnisse führen kann. Durch die weiterentwickelte Methode hingegen konnten diese Probleme dank Verzichts auf klassische Matrixsubstanzen eliminiert werden mit dem zusätzlichen Vorteil, nun auch UV-absorbierende Moleküle direkt auf der Probenoberfläche zu desorbieren, ionisieren und nachfolgend messen zu können. In Gewebeproben von Hypericum perforatum (Echtes Johanniskraut) und Blütenblättern von Arabidopsis thaliana (Ackerschmalwand) konnte mit dieser Methode beispielsweise eine ungewöhnliche Verteilung von phenolischen Verbindungen sichtbar gemacht werden (Abb. 3a): Der obere Teil eines Blütenblattes von Arabidopsis thaliana war angereichert mit Kaempferol, Kaempferol-Rhamnosid und weiteren Kaempferol-Glycosiden mit höherem Molekulargewicht, dagegen enthielt der untere Teil des Blütenblattes eher stärker oxidierte Kaempherol-Derivate wie Quercetin, Isorhamnetin und weitere Glycoside dieser Verbindungen [6].
In einem aktuellen Projekt wird versucht, die Verteilung von Kohlenwasserstoffen und anderen unpolaren Lipiden (Glycerol und Fettalkoholester) in Fruchtfliegen, insbesondere Drosophila melanogaster, abzubilden. Die minimalen Ausmaße und die Dreidimensionalität des winzigen Insekts machen das Projekt zu einer großen Herausforderung. Bislang konnten die mehr oder weniger ebenflächigen Flügel erfolgreich analysiert werden: Abgebildet werden konnten Kohlenwasserstoff-Monoene und -Diene, also vermeintliche Sexualpheromone, sowie verschiedene Wachsester.
Vielen Laboren, die routinemäßig mit MALDI-TOF/MS-Instrumenten arbeiten, ist die LDI-Imaging-Technik zu empfehlen: sie ist einfach und die Probenvorbereitung sowie nachfolgende Arbeiten sind weniger aufwändig.
Zukunftsperspektiven
Die Auswahl und Applikation der Matrixsubstanz scheint der limitierende Faktor beim bildgebenden Verfahren der Massenspektrometrie zu sein. Herkömmliche Matrices sind für die Analyse kleiner Metaboliten nicht brauchbar, denn deren Signale werden von einem dichten Wald Matrix-bezogener Peaks überlagert. Basierend auf den klassischen Prinzipien der Säure-Base-Chemie konnten wir kürzlich eine neue Klasse von „ionenfreien“ Matrices entwickeln [8] und mithilfe von Erkenntnissen aus der theoretischen Chemie die kritischen chemischen Eigenschaften bestimmen, die für ihre Auswahl und ihr rationales Design maßgeblich sind. Derzeit werden neue Protokolle für das Imaging von Säuren in verschiedenen Geweben unter Einsatz dieser neuen Klasse von Matrices erstellt.
Alle beschriebenen Experimente müssen immer noch unter Hochvakuumbedingungen durchgeführt werden, was aber nicht der natürlichen Umgebung einer biologischen Probe entspricht. Aktuelle Pläne sind daher auf ein Imaging unter natürlichen Außenbedingungen gerichtet. Unser Ziel ist es, zeitliche und räumliche Muster wichtiger chemischer Signale von Bakterien, Insekten oder Pflanzen möglichst unbeeinflusst von der Messtechnik zu ermitteln. Auf diese Weise hoffen wir, die noch verborgenen Details der chemischen Kommunikation zwischen Organismen aufzudecken, wie zum Beispiel die Aufklärung der Chemie zwischen Männchen und Weibchen vor der Paarung.
In naher Zukunft ist auch die Entwicklung neuer Methoden für metabolomische Untersuchungen und das Imaging einzelner Zellen geplant. Die Möglichkeit, metabolomische Analysen auf die Ebene einzelner Zellen zu bringen, sollte die Beantwortung vieler offener Fragen in Bezug auf die Signalübermittlung von Zelle zu Zelle ermöglichen – zum Beispiel die Frage nach der Ausbreitung schneller Oxidationsreaktionen (ROS) von Zelle zu Zelle, die bislang aufgrund des Mangels an angemessenen molekular-selektiven Werkzeugen nicht untersucht werden konnte. Deren Komponenten könnten mithilfe neuer metabolomischer Methoden auf der Ebene einzelner Zellen, die derzeit in vielen Laboren intensiv erarbeitet werden, visualisiert werden [9].

![Massenspektrometrisches Imaging eines Blattes der Ackerschmalwand (Arabidopsis thaliana)(a) Ausschnitt (m/z 400 - 500) eines MALDI-TOF/MS-Spektrums, gemittelt aus 100 aufeinanderfolgenden Pixeln auf einem Blatt, das auf der Blattunterseite mit 9-Aminoacridin als Matrix besprüht wurde. Die Peaks bei 436,0, 447,0 und 492,0 m/z entsprechen den M--Ionen von 4-Methylsulfinylbutylglucosinolat (4MSOB), Indol-3-ylmethylglucosinolat (I3M) und 8-Methylsulfinyloctylglucosinolat (8MSOO). Die Spektren wurden in einem Negative-reflectron-Modus auf einem MALDI micro MX (Waters, Manchester, UK) erfasst. (b) 3D-Karte der Ionenintensität des Matrix-Ions (m/z 193,0 ± 0,25 [M - H]-) resultierend aus der Messung einer Blattprobe, die auf einem MALDI-Objektträger aufgebracht worden war. Das Bild zeigt, dass die Matrix gleichmäßig über das gesamte Blatt aufgetragen wurde; dazu wurde die zu besprühende Fläche mit einer Papiermaske festgelegt, um ein Streuen der Matrix während der Anwendung zu verhindern. (c) Ionenintensitätskarte von 4MSOB (m/z 436,0 ± 0.25), die mit Hilfe der ImageJ-Software aus rund 100.000 MALDI-TOF/MS-Spektren erstellt wurde (420 × 252 Pixel (Breite × Höhe), Pixelgröße 200 μm). Die Ionenintensitäten sind in Falschfarbe (rot) auf einer Intensitätsskala von 0-255 Farbtiefen dargestellt. (d) Überlagerung des Fraßbildes der Raupe Helicoverpa armigera (Baumwoll-Kapseleule) und der MSI-Verteilungskarte des Giftstoffes Methylsulfinylbutylglucosinolat (4MSOB; es wurde nicht dasselbe Blatt verwendet). Massenspektrometrisches Imaging eines Blattes der Ackerschmalwand (Arabidopsis thaliana)(a) Ausschnitt (m/z 400 - 500) eines MALDI-TOF/MS-Spektrums, g](https://www.mpg.de/1191802/original-1345135487.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE5MTgwMn0%3D--c99f3ba111fe479f5171196b06e54cd3b0280833)
![Laser-Desorption/Ionisierung (LDI-Imaging).(a) Abbildung eines einzelnen Blütenblattes der Ackerschmalwand (Arabidopsis thaliana, i) sowie im Negative-ion-Modus erstellte LDI-MSI-Images zur Darstellung der Verteilung von 285 m/z (ii, entspricht Kaempferol) und 301 m/z (iii, entspricht Quercetin). Die unterschiedliche Verteilung der beiden Stoffwechselprodukte, die sich nur in ihrem Oxidationsgrad unterscheiden, ist deutlich zu erkennen. Die Strukturformeln sind unten links und Mitte dargestellt. (b) Ausgewählte LDI-TOF/MS-Aufnahme einer antibiotischen Substanz. Gezeigt ist die äußere (in der Abbildung oben) und die innere Seite (in der Abbildung unten) eines Bienenwolfkokons. (i) Ionenintensitätskarten von Streptochlorin [m/z 219 ± 0.5 [M+H] +]; die Ionenintensitäten der jeweiligen Messpunkte sind Heatmap-kodiert, wobei die schwarze Farbe 0 Zählungen und die rote Farbe 255 Zählungen entspricht. (ii) Abbildung des Kokons, der für das LDI-TOF/MS-Imaging auf einem MALDI-Objektträger befestigt wurde (Skalierbalken: 5 mm). Laser-Desorption/Ionisierung (LDI-Imaging).(a) Abbildung eines einzelnen Blütenblattes der Ackerschmalwand (Arabidopsis thaliana, i) sowie im Negati](https://www.mpg.de/1191895/original-1345135488.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE5MTg5NX0%3D--f355e1d835f5a39099d072bbc20bbb1f83ac8fe5)